R のパッケージ e1071 は、libsvm へのインターフェースを提供している。libsvm アルゴリズムで予測機を簡単に作成できる。
e1071パッケージの使い方
e1071 パッケージの svm 関数で簡単にモデルの作成ができる。
library(e1071)
data(iris)
# libsvm でモデル作成
model <- svm(
Species ~ ., data = iris, # irisのSpeciesを他の特徴量で予測
method = "C-classification", # 分類方法
kernel = "linear", # カーネル関数
cost = 2 # Cパラメーター
)
# モデルの確認
model
## Call:
## svm(formula = Species ~ ., data = iris, method = "C-classification",
## kernel = "linear", cost = 2)
##
## Parameters:
## SVM-Type: C-classification
## SVM-Kernel: linear
## cost: 2
## gamma: 0.25
##
## Number of Support Vectors: 24
# 予測用にサンプルデータを5つ作成します
unknown <- data.frame(
Sepal.Length = c(5.1, 5.2, 6.7, 4.5, 7.0),
Sepal.Width = c(3.5, 3.3, 4.1, 3.1, 5.0),
Petal.Length = c(1.1, 1.5, 2.1, 6.7, 4.1),
Petal.Width = c(0.2, 0.1, 0.3, 1.2, 1.4)
)
# 予測結果
predict(model, unknown, decision.values = TRUE)
## 1 2 3 4 5
## setosa setosa setosa virginica versicolor
## attr(,"decision.values")
## setosa/versicolor setosa/virginica versicolor/virginica
## 1 1.8208103 1.47264811 12.167505
## 2 1.5335990 1.34830896 11.547934
## 3 0.7999217 1.15153503 11.701613
## 4 -2.2194464 -1.10219176 -2.061327
## 5 -1.0224676 -0.01898811 5.856400
## Levels: setosa versicolor virginica交差検定
次のように cross 引数にデータの分割数を指定することで、簡単に交差検定を行うことができる。
model <- svm(Species ~ ., data = iris, cross = 5)
# モデルの確認
summary(model)
## Call:
## svm(formula = Species ~ ., data = iris, cross = 5)
##
## Parameters:
## SVM-Type: C-classification
## SVM-Kernel: radial
## cost: 1
## gamma: 0.25
##
## Number of Support Vectors: 51
## ( 8 22 21 )
##
## Number of Classes: 3
##
## Levels:
## setosa versicolor virginica
##
## 5-fold cross-validation on training data:
##
## Total Accuracy: 94.66667
## Single Accuracies:
## 93.33333 93.33333 93.33333 96.66667 96.66667 パラメーターの調整
R のパッケージに搭載されている iris のデータを利用してパラメーター調整を行う方法として、もっとも簡単な方法はグリッドサーチである。これには tune.svm 関数を利用する。
library(e1071)
data(iris)
# グリッドサーチの範囲
gamma.lim <- 10^(seq(-5, 5, 0.1))
cost.lim <- 10^(seq(-1, 1, 0.1))
tune <- tune.svm(
Species ~ ., data = iris,
gamma = gamma.lim, # gammaパラメーターをgamma.limの範囲で移動させる
cost = cost.lim, # costパラメーターをcost.limの範囲で移動させる
tunecontrol = tune.control(sampling = "cross", cross = 5) # 5-cross validation
)
# グリッドサーチの結果を確認
tune
## Parameter tuning of ‘svm’:
##
## - sampling method: 5-fold cross validation
##
## - best parameters:
## gamma cost
## 0.1584893 1
##
## - best performance: 0.02666667結果から gamma = 0.1585、cost = 1 が最適パラメーターであることがわかります。
グリッドサーチの結果を plot することで、gamma と cost の二次元プロットが見られます。
plot(tune)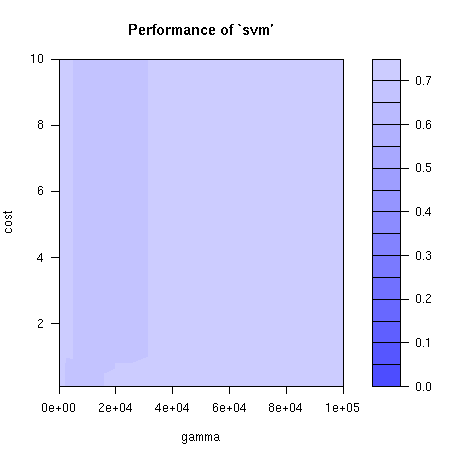
References
- Estimating the support of a high-dimensional distribution. Neural Computation 2001, 13:1443-71. ACM
- Misc Functions of the Department of Statistics (e1071) TU Wien 2005.